.
Manter o controle de vários fusos horários torna-se crucial quando você realiza reuniões de negócios internacionais, viaja para diferentes partes do mundo ou se comunica com amigos e familiares que moram no exterior. Calcular manualmente as diferenças de horário entre duas cidades pode ser agitado e complicado.
Felizmente, o seu iPhone oferece maneiras convenientes de visualizar vários fusos horários, facilitando a sincronização com o resto do mundo. Abaixo, orientaremos você através de três métodos de exibição de vários fusos horários no seu iPhone usando o aplicativo Relógio e widgets iOS.
Como verificar vários fusos horários no aplicativo Relógio
Você pode usar o aplicativo Relógio para controlar facilmente vários fusos horários no seu iPhone. Essa abordagem é a maneira mais simples e conveniente de fazer isso.
Abra o aplicativo Relógio e vá para o Relógio mundial guia no menu inferior. Toque em mais (+) botão no canto superior direito da tela. Isso abre um pop-up onde você pode pesquisar e selecionar a cidade para a qual deseja ver a hora.
Agora você poderá ver a hora na cidade selecionada no Relógio mundial e o número de horas que está atrasado ou adiantado em relação à hora local. Você pode repetir esse processo para várias cidades e fusos horários.
A única desvantagem desse método é que você precisa abrir o aplicativo Relógio sempre que desejar ver a hora. Funciona melhor apenas se você quiser visualizar fusos horários de diferentes regiões ocasionalmente e não regularmente.
Como adicionar vários fusos horários à tela inicial do seu iPhone
Se você precisa verificar frequentemente os fusos horários de várias regiões, uma opção conveniente é exibir vários relógios na tela inicial do seu iPhone. Você pode fazer isso usando o widget Relógio Mundial. Para adicionar este widget à sua tela inicial, siga estas etapas:
- Mantenha pressionado um espaço vazio na tela inicial até que os aplicativos comecem a balançar.
- Toque em mais (+) botão no canto superior esquerdo. Em seguida, role para baixo e toque em Relógio.

- Deslize para a esquerda várias vezes para acessar Relógio mundial ferramenta. Este widget vem em duas opções de estilo – quadrado e retangular – e inclui quatro relógios exibindo os horários de quatro cidades diferentes.
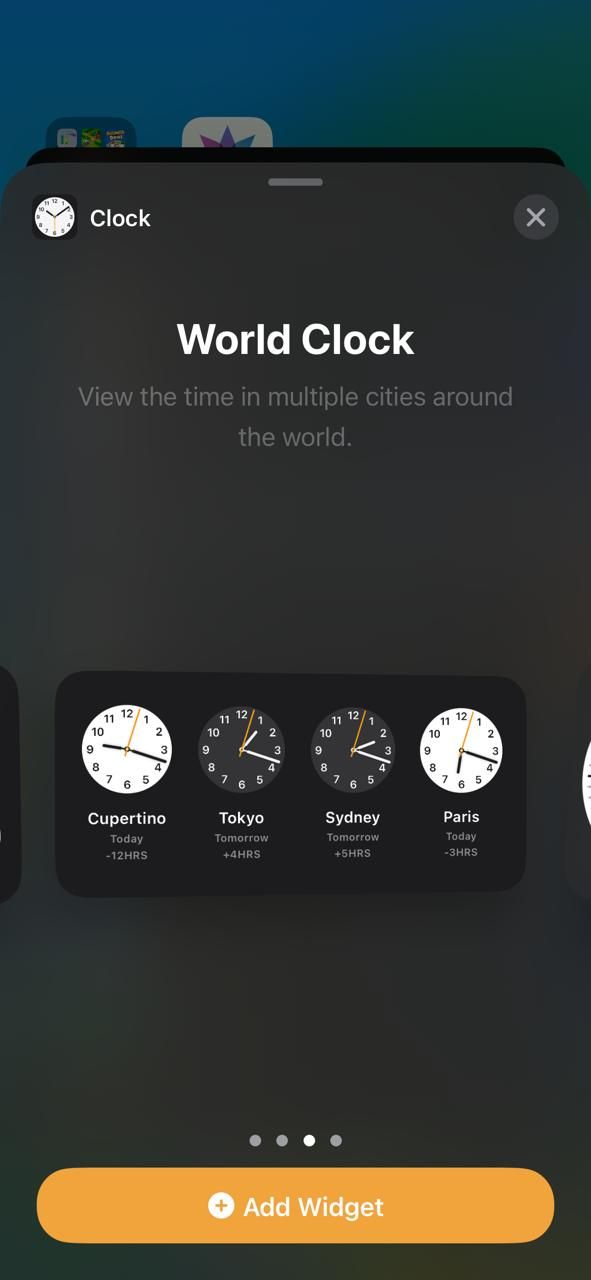
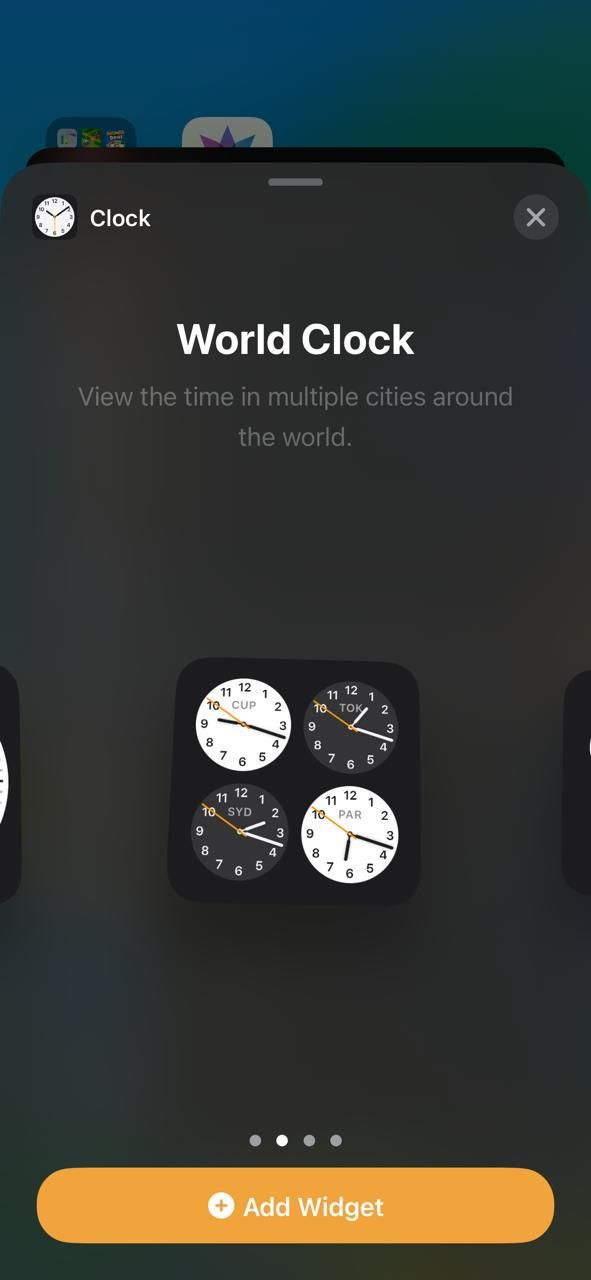
- Escolha seu estilo preferido e toque em Adicionar widget na parte inferior para adicioná-lo à tela inicial. Você verá quatro cidades aleatórias no widget.
- Agora, mantenha pressionado o widget que você adicionou e toque em Editar widget. Toque na cidade que deseja alterar e selecione a cidade desejada. Repita isso para todos os quatro relógios.
Você finalmente configurou o widget Relógio Mundial com as cidades que você gosta. Também recomendamos experimentar alguns desses aplicativos incríveis que permitem criar widgets personalizados para o seu iPhone para obter mais opções de personalização.
Como adicionar vários fusos horários à tela de bloqueio do seu iPhone
Para uma acessibilidade ainda maior, você também pode exibir fusos horários de várias cidades na tela de bloqueio do seu iPhone. Existem várias opções de widget de tela de bloqueio que você pode escolher.
Mantenha pressionado a tela de bloqueio do seu iPhone e toque no Customizar opção na parte inferior. Em seguida, selecione Tela de bloqueio e depois toque Adicionar widgets > Relógio.
Você verá várias opções de widgets para o aplicativo Relógio:
- Cidade Digital: Este widget permite adicionar um relógio digital de uma cidade específica para verificar a hora naquele local. Você pode adicionar até quatro relógios digitais pequenos ou dois relógios retangulares usando esta opção.
- Análogo da cidade: Este widget também permite exibir até quatro pequenos relógios analógicos para vários fusos horários.
- Relógio mundial: Este widget permite visualizar a hora em três cidades ao redor do mundo.
Toque no seu widget preferido para adicioná-lo à tela de bloqueio e, em seguida, toque no widget adicionado para selecionar as cidades para as quais deseja exibir a hora.
Para o widget Relógio mundial, toque em cada cidade individual para alterá-la para a cidade desejada. Quando estiver satisfeito com sua escolha, toque em Feito no canto superior direito da tela.
Visualize facilmente vários fusos horários no seu iPhone
Quer você seja um viajante frequente, um profissional que lida com clientes globais ou simplesmente alguém que deseja ficar conectado com amigos e familiares em todo o mundo, seu iPhone oferece várias opções para ajudá-lo a visualizar vários fusos horários.
O aplicativo Relógio é suficiente se você não precisa verificar a hora em diferentes cidades com frequência. No entanto, se você precisa verificar regularmente os fusos horários em diferentes regiões do mundo, é uma boa ideia considerar adicionar widgets de relógio à tela inicial ou à tela de bloqueio.
.








