.

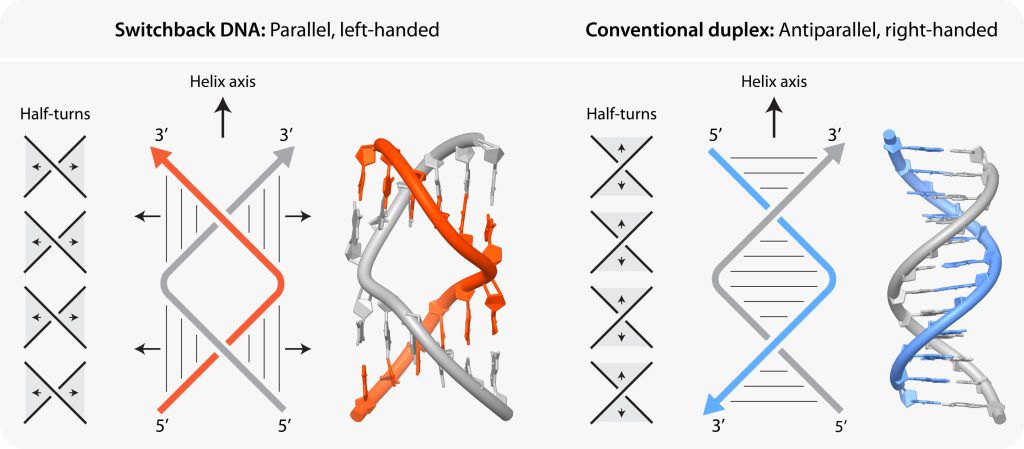
O DNA Switchback é composto de seções estruturais chamadas “meias voltas” que se assemelham ao DNA normal, mas são organizadas de forma diferente. Este diagrama demonstra essas diferenças estruturais, que dão ao DNA Switchback sua capacidade única de se dobrar, entre outras propriedades distintas. Crédito: Comunicações da Natureza (2024). DOI: 10.1038/s41467-024-50348-3
Ao descobrir novas maneiras de manipular a matéria nos níveis atômico e molecular, os avanços na nanotecnologia estão abrindo caminho para inovações na medicina, eletrônica, ciência dos materiais e recuperação ambiental, entre muitas outras áreas.
Uma especialidade importante neste campo — e uma área de estudo de destaque no Instituto de RNA da Universidade de Albany — é a nanotecnologia de DNA, na qual os pares de bases que compõem as moléculas de DNA são manipulados para construir pequenas estruturas em diferentes formatos que podem ser usadas para aplicações que incluem administração de medicamentos, diagnósticos médicos e até mesmo armazenamento de dados.
Pesquisadores do Instituto RNA, incluindo o bolsista de pós-doutorado Bharath Raj Madhanagopal e o cientista pesquisador sênior Arun Richard Chandrasekaran, juntamente com uma equipe de colaboradores da UAlbany, foram coautores de um novo estudo que explorou as propriedades únicas de um certo tipo de nanoestrutura de DNA chamada “DNA ziguezague”, que pode ter implicações no dobramento do DNA na natureza e ser útil no desenvolvimento de novos tipos de nanoestruturas com aplicações biomédicas.
As suas descobertas foram publicadas em Comunicações da Natureza.
Aqui, Madhanagopal e Chandrasekaran compartilham insights sobre os fundamentos de seu campo e os avanços que aguardam com novas descobertas na nanotecnologia de DNA.
O que são nanoestruturas de DNA e por que elas são importantes?
Muitos conhecem o DNA como a molécula que armazena a informação genética que é passada de uma geração para a outra. As propriedades químicas do DNA que o tornam uma molécula excelente para armazenar informação genética também o tornam um material de construção útil — especialmente quando se trata de fazer objetos minúsculos, tão pequenos quanto alguns nanômetros.
As sequências das quatro nucleobases no DNA — adenina, guanina, timina e citosina — são inerentemente programáveis. Isso ocorre porque a adenina sempre pareia com a timina, e a guanina com a citosina. Esses padrões confiáveis no pareamento de bases nos permitem projetar cadeias específicas de DNA que se unem como blocos de Lego para formar nanoestruturas.
Ao usar DNA para construir nanoestruturas, podemos atingir excelente precisão no tamanho das estruturas. Também podemos fazer objetos de diversas formas e complexidades arquitetônicas — capacidades que não são facilmente alcançadas usando outras tecnologias. Nanoestruturas de DNA estão sendo desenvolvidas para uso em administração de medicamentos, diagnósticos e armazenamento de dados, para citar algumas aplicações.
O que é ‘DNA switchback’?
Assim como usamos tijolos para construir prédios, usamos blocos de construção de tamanho nanométrico chamados “motivos” e “ladrilhos” feitos de DNA para criar estruturas elaboradas em nanotecnologia de DNA. Semelhante a como os tijolos podem vir em diferentes formas e tamanhos, o mesmo pode acontecer com motivos e ladrilhos. Criar esses motivos estruturais e entender suas propriedades é a base da pesquisa em nanotecnologia de DNA.
“Switchback DNA” é um dos primeiros motivos de DNA projetados por Nadrian Seeman, o fundador do campo da nanotecnologia de DNA. Queríamos explorar como suas curiosas características estruturais se manifestariam em nanoestruturas. Ao estudar as propriedades do switchback DNA, acreditamos que podemos criar nanoobjetos baseados em DNA ainda mais diversos com propriedades exóticas.
O que torna o DNA Switchback único?
O DNA Switchback tem apenas duas fitas, então ele pode ser comparado diretamente com a estrutura de dupla hélice do DNA com a qual todos estão familiarizados. No DNA switchback, as duas fitas têm seções, chamadas meias-voltas, que lembram o DNA normal, mas a maneira como elas são organizadas torna o DNA switchback único.
Normalmente, o DNA é uma hélice dupla com sentido helicoidal destro por toda a molécula. No DNA switchback, meias voltas destras são organizadas de tal forma que a molécula como um todo é uma hélice dupla canhota. Isso ocorre porque se você traçar a espinha dorsal do DNA ao longo da hélice, verá que, após cada meia volta, os fios se dobram para trás. Essas diferenças são ilustradas no diagrama abaixo.
Descobrimos que a estrutura única do DNA switchback pode afetar propriedades importantes para seu papel potencial em aplicações biomédicas — coisas como estabilidade estrutural, vulnerabilidade a enzimas e propriedades imunogênicas, que, por exemplo, podem impactar a capacidade de uma nanoestrutura de efetivamente entregar um medicamento a um tecido específico. Entender essas propriedades e descobrir quais podem ser controladas e como controlá-las é essencial.
O que o campo ganha ao entender melhor o DNA do switchback?
Os resultados deste estudo ajudarão os pesquisadores que produzem nanoestruturas de DNA a melhorar seus projetos usando blocos de construção de DNA em formato de zigue-zague.
Por exemplo, agora sabemos que uma enzima comum chamada “DNase I” não degrada o DNA switchback tão rapidamente quanto degrada o B-DNA convencional (o DNA que geralmente é encontrado em organismos vivos). Se quisermos usar nanoestruturas de DNA para transportar medicamentos para os tecidos do corpo, não queremos que uma enzima quebre a nanoestrutura antes que ela possa atingir o tecido-alvo.
Se isso acontecesse, o medicamento não seria eficaz. Agora podemos considerar incorporar o DNA switchback para ajudar a mitigar esse desafio, que é um obstáculo comum no campo.
Também descobrimos que há sequências genéticas no genoma humano que podem potencialmente se dobrar em DNA switchback. Nossos resultados sugerem que, sob algumas condições, o DNA com padrões de repetição específicos pode formar DNA switchback. Essas sequências são prevalentes nos cromossomos de animais e plantas, e podem adotar formas estruturais sobre as quais sabemos muito pouco.
É emocionante saber que essas sequências podem se dobrar em DNA switchback em um tubo de ensaio sob certas condições. Se isso pode acontecer em uma célula viva ainda está para ser visto.
Como essas sequências repetitivas estão envolvidas em doenças como distrofia miotônica e doença de Huntington, essa via de estudo pode nos ajudar a entender melhor essa classe de doenças e também pode nos ajudar a descobrir novos alvos de medicamentos para essas doenças no futuro.
Quais são os maiores aprendizados deste trabalho?
Nosso trabalho com o DNA switchback mostra que podemos “ajustar” as propriedades do DNA dobrando-o em diferentes padrões sem modificações químicas. Entender as propriedades do DNA switchback será útil na criação de dispositivos de DNA para biossensores, administração de medicamentos, computação de DNA e outras aplicações.
Nossas descobertas também demonstram que as regras de complementaridade que Watson e Crick definiram em seu icônico modelo de dupla hélice da estrutura do DNA precisam ser expandidas. No modelo proposto por Watson e Crick, as direções das duas fitas são opostas. Isso significa que uma extremidade da primeira fita interage com a extremidade oposta da segunda fita.
No DNA switchback, o padrão de pareamento de bases é diferente. Enquanto a maioria das regras de complementaridade definidas por Watson e Crick se aplicam ao DNA switchback, a posição dos pares de bases difere.
Finalmente, nossa hipótese de que sequências repetidas poderiam formar estruturas de DNA em zigue-zague abre discussões interessantes — e estudos futuros — sobre a ocorrência biológica dessas estruturas de DNA não tradicionais.
Mais Informações:
Bharath Raj Madhanagopal et al, As propriedades estruturais incomuns e a relevância biológica potencial do DNA switchback, Comunicações da Natureza (2024). DOI: 10.1038/s41467-024-50348-3
Fornecido pela Universidade de Albany
Citação: O formato das moléculas que virão: Perguntas e respostas sobre o design de nanoestruturas de DNA para aplicações biomédicas (2024, 6 de agosto) recuperado em 6 de agosto de 2024 de https://phys.org/news/2024-08-molecules-qa-dna-nanostructures-biomedical.html
Este documento está sujeito a direitos autorais. Além de qualquer uso justo para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem permissão por escrito. O conteúdo é fornecido apenas para fins informativos.
.