.
Dinâmica conformacional do vRNP da influenza A. Crédito: 2024 Carlero, et al., American Chemical Society
Pesquisadores do Nano Life Science Institute (WPI-NanoLSI), da Universidade de Kanazawa, do IMDEA Nanoscience (Madrid, Espanha) e do CNB-CSIC (Madrid, Espanha) relatam em ACS Nano experimentos que revelam um ciclo de estágios conformacionais pelos quais os genomas recombinantes da gripe A passam durante a síntese de RNA.
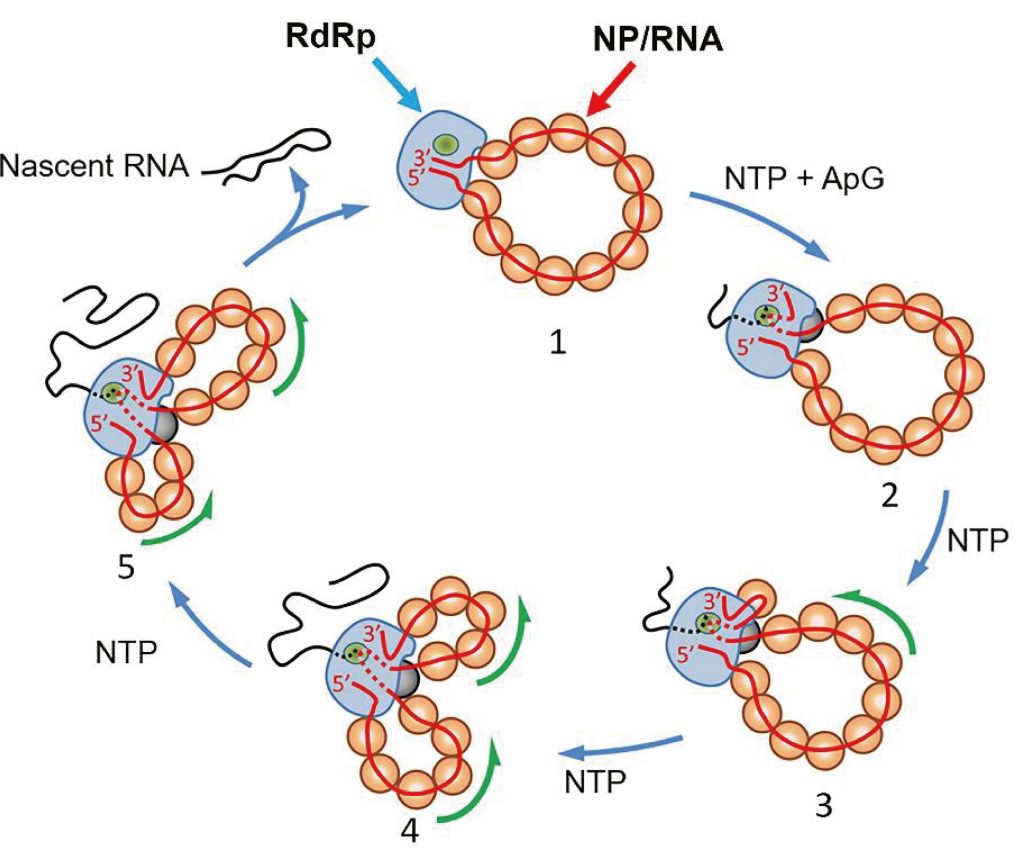
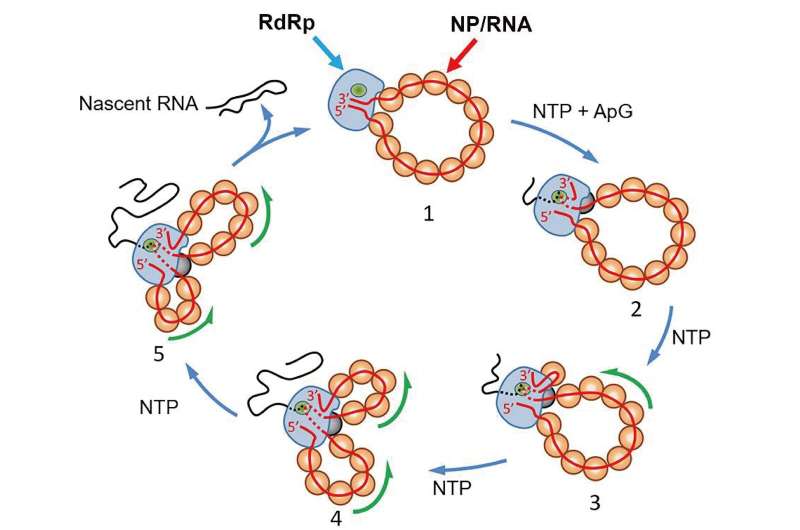
A influenza A é um risco global à saúde responsável por epidemias locais e pandemias mortais. Como tal, o mecanismo pelo qual o vírus se replica atraiu interesse significativo. Pesquisadores liderados por Shingo Fukuda no Nano Life Science Institute (WPI-NanoLSI) na Universidade de Kanazawa no Japão, Jaime Martin-Benito no CNB-CSIC e Borja Ibarra no IMDEA Nanociencia na Espanha, usaram microscopia de força atômica de alta velocidade e microscopia eletrônica para fixar a dinâmica conformacional de genomas virais recombinantes (ou rRNPs) durante a síntese de RNA.
Tentativas anteriores de entender quais possíveis mudanças conformacionais ocorrem durante o ciclo de multiplicação viral da gripe A foram dificultadas pelo que os pesquisadores descrevem como a “estrutura dupla-helicoidal volumosa” dos RNPs virais (vRNP), o que dificultava a visualização do que estava acontecendo.
Como resultado, os pesquisadores produziram um complexo circular de ribonucleoproteína recombinante (rRNP), o que lhes permitiu superar o “problema volumoso”. Os autores usaram HS-AFM para acompanhar mudanças conformacionais de complexos rRNP individuais em tempo real durante a síntese ativa de RNA.
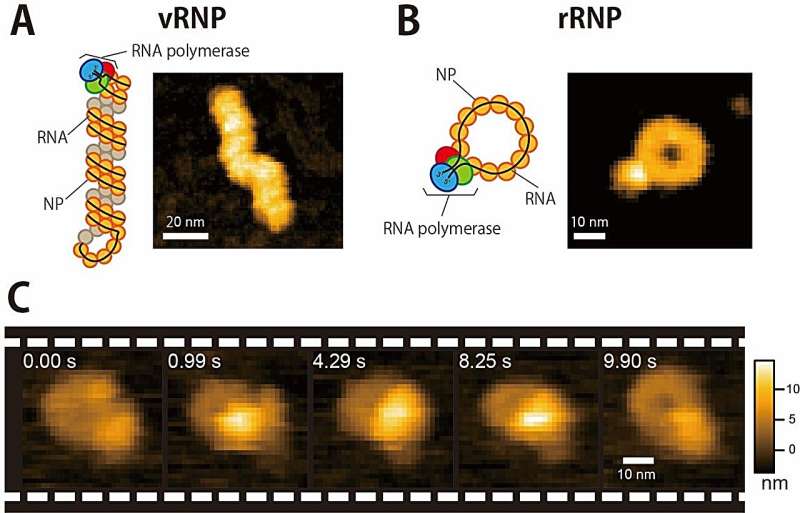
(A, B) Imagens HS-AFM de vRNP (A) e rRNP (B). (C) Imagens HS-AFM sucessivas de rRNP durante a síntese de RNA. Crédito: 2024 Carlero, et al., American Chemical Society
O trabalho deles fornece a primeira evidência experimental direta mostrando que rRNPs individuais podem ser reciclados para múltiplos ciclos de transcrição e replicação. Esta é uma característica fundamental para a multiplicação viral. Além disso, o estudo deles destaca como fatores que afetam a estabilidade das estruturas secundárias do RNA nascente afetam a taxa de síntese de RNA.
Os autores concluíram que a abordagem é útil para investigar mecanismos de transcrição e replicação virais. “A pausa transcricional é uma propriedade intrínseca da maioria das RNA polimerases, e sua regulação constitui um dos mecanismos centrais de controle da expressão gênica”, acrescentam.
“Futuros experimentos de molécula única de cinética de síntese de RNA em tempo real pelo IAV RdRp [influenza A virus RNA dependent RNA polymerase] dentro do contexto do RNP ajudará a elucidar a natureza dos estados de pausa putativos e seus papéis na transcrição e replicação viral.”
Mais Informações:
Diego Carlero et al, Dinâmica conformacional dos complexos de ribonucleoproteína do vírus influenza A durante a síntese de RNA, ACS Nano (2024). DOI: 10.1021/acsnano.4c01362
Fornecido pela Universidade de Kanazawa
Citação: Estudos de microscopia de força atômica de alta velocidade fornecem insights sobre a replicação viral da gripe A (2024, 5 de agosto) recuperado em 5 de agosto de 2024 de https://phys.org/news/2024-08-high-atomic-microscopy-insights-influenza.html
Este documento está sujeito a direitos autorais. Além de qualquer uso justo para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem permissão por escrito. O conteúdo é fornecido apenas para fins informativos.
.