.
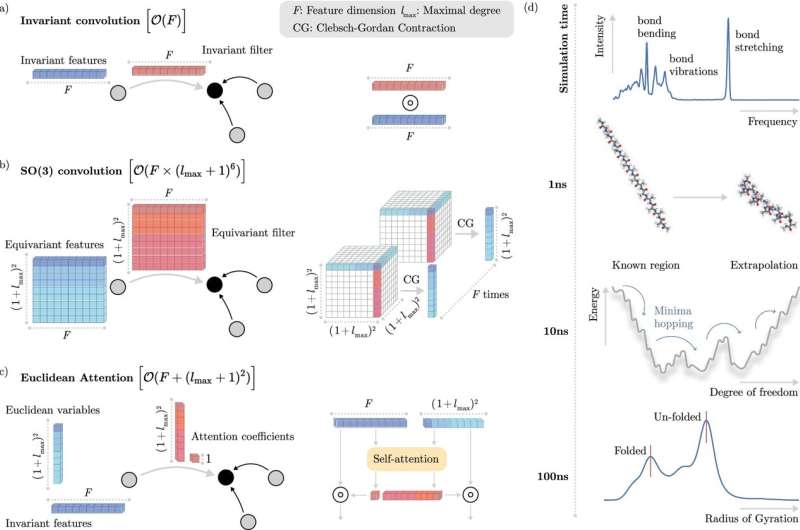
Visão geral dos resultados. Crédito: Comunicações da Natureza (2024). DOI: 10.1038/s41467-024-50620-6
Átomos são sistemas quânticos complexos que consistem em um núcleo carregado positivamente cercado por elétrons carregados negativamente. Quando múltiplos átomos se juntam para formar uma molécula, os elétrons dos átomos constituintes interagem de uma maneira complicada, tornando a simulação computacional de moléculas um dos problemas mais difíceis da ciência moderna.
Pesquisadores do Instituto de Berlim para Fundamentos de Aprendizagem e Dados (BIFOLD) da TU Berlin e do Google DeepMind desenvolveram um novo algoritmo de aprendizado de máquina que permite simulações altamente precisas da dinâmica de uma única ou múltiplas moléculas em longas escalas de tempo. O trabalho deles foi publicado em Comunicações da Natureza.
Essas chamadas simulações de dinâmica molecular são importantes para entender as propriedades de moléculas e materiais e têm aplicações potenciais no desenvolvimento de medicamentos e design de materiais (por exemplo, para uso em painéis solares e baterias). Métodos tradicionais para calcular as interações de elétrons dependem de encontrar soluções da chamada equação de Schrödinger.
A equação de Schrödinger descreve os níveis de energia que um sistema quântico — por exemplo, átomos ou moléculas — pode assumir. Esta é uma tarefa notoriamente difícil, e encontrar uma solução para moléculas contendo mais do que algumas dezenas de átomos pode levar vários dias — mesmo em computadores poderosos. Para piorar a situação, para executar simulações de dinâmica molecular em longas escalas de tempo, a equação de Schrödinger precisa ser resolvida milhares ou até milhões de vezes, fazendo com que o custo computacional exceda rapidamente os recursos de computação disponíveis hoje.
“A simulação dessas interações e as previsões resultantes para processos complexos como o dobramento de proteínas ou a ligação entre moléculas individuais é um sonho antigo de muitos químicos e cientistas de materiais, e economizaria muitos experimentos caros e trabalhosos”, explica o pesquisador do BIFOLD, Thorben Frank.
Nos últimos anos, métodos de machine learning (ML) trouxeram esse sonho ao alcance. Em vez de resolver explicitamente a equação de Schrödinger, eles podem aprender a prever diretamente o resultado geral das interações eletrônicas relevantes no nível atomístico, com custo computacional bastante reduzido.
A dificuldade então é deslocada para encontrar algoritmos eficientes para “ensinar” ao sistema de aprendizado de máquina como os elétrons interagem sem modelá-los explicitamente. Para reduzir a complexidade dessa tarefa, muitos algoritmos de aprendizado usam o fato de que sistemas físicos seguem as chamadas invariâncias.
Simplificando, certas propriedades das moléculas permanecem as mesmas quando as moléculas são movidas no espaço, mas as distâncias relativas entre átomos individuais permanecem fixas — o que significa que a máquina não precisa aprender nada de novo nesses casos. No entanto, a maneira como essas invariâncias são tipicamente incorporadas em modelos de ML é computacionalmente cara, limitando, em última análise, a velocidade com que os modelos podem executar simulações de dinâmica molecular.
Para lidar com essa deficiência, os cientistas do BIFOLD criaram um novo algoritmo de aprendizado que desacopla invariâncias de outras informações sobre um sistema químico desde o início. Ao contrário dos métodos anteriores que exigiam a extração de componentes invariantes de cada operação dentro do modelo, essa nova abordagem simplifica o processo. Agora, o modelo ML pode reservar as operações mais complexas para as informações físicas que realmente importam e reduzir drasticamente o custo computacional geral.
“Simulações que exigiam meses ou até anos de computação em clusters de computadores de alto desempenho, agora podem ser realizadas em poucos dias em um único nó de computador. O salto em eficiência permite simulações de escala de longo prazo, que são necessárias para entender a estrutura, a dinâmica e o funcionamento de sistemas atomísticos. Isso permite, portanto, insights mais profundos sobre os processos mais complexos e fundamentais da natureza”, diz o pesquisador do BIFOLD, Dr. Stefan Chmiela, que liderou o projeto de pesquisa.
No futuro, a simulação precisa da interação de moléculas com proteínas no corpo humano poderá permitir que os pesquisadores desenvolvam novos medicamentos sem a necessidade de realizar experimentos, economizando tempo e dinheiro e, ao mesmo tempo, sendo mais ecológico.
Para mostrar potenciais aplicações do algoritmo, a equipe usou o novo método ML para identificar a versão mais estável do ácido docosahexaenoico, um ácido graxo que é um componente estrutural primário no cérebro humano. Essa tarefa requer a varredura de dezenas de milhares de candidatos potenciais com alta precisão. Até agora, tal análise teria sido inviável com métodos mecânicos quânticos tradicionais.
Conforme observado pelo Prof. Dr. Klaus-Robert Müller, codiretor do BIFOLD e cientista principal do Google DeepMind, “Este trabalho demonstra o potencial de combinar técnicas avançadas de aprendizado de máquina com princípios físicos para superar desafios de longa data na química computacional. Ele continua uma linha crítica de pesquisa que coloca o foco em escalonar abordagens de ML para sistemas químicos realistas de interesse prático.”
O Dr. Oliver Unke, cientista pesquisador sênior do Google DeepMind, comenta: “No início deste ano, conseguimos dimensionar modelos para milhares de átomos, mas com novos avanços como esse, pode ser possível migrar para números ainda maiores de átomos.”
Enquanto simulações com dezenas a centenas de milhares de átomos estão se tornando acessíveis agora, algumas estruturas consistem em milhões de átomos ou mais. A próxima geração de algoritmos precisará ser capaz de simular tais tamanhos de sistema com precisão, o que requer uma descrição correta de interações físicas adicionais, complexas e de longo alcance.
Mais Informações:
J. Thorben Frank et al, Um transformador euclidiano para campos de força aprendidos por máquina rápidos e estáveis, Comunicações da Natureza (2024). DOI: 10.1038/s41467-024-50620-6
Fornecido pela Universidade Técnica de Berlim
Citação: Calculando mais rápido: Acoplando IA com física fundamental (2024, 6 de agosto) recuperado em 6 de agosto de 2024 de https://phys.org/news/2024-08-faster-coupling-ai-fundamental-physics.html
Este documento está sujeito a direitos autorais. Além de qualquer uso justo para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem permissão por escrito. O conteúdo é fornecido apenas para fins informativos.
.